Aux côtés de Sorbonne Université, la deeptech française Qubit Pharmaceuticals a développé un modèle d’intelligence artificielle destiné à la simulation moléculaire : FeNNix-Bio1. Un modèle dit « de fondation », à la précision et à la rapidité sans pareil d’après ses créateurs, qui viennent de lever le voile, au travers de deux publications en cours d’évaluation par les pairs, sur ce qui s’annonce comme un véritable couteau suisse de la modélisation en chimie.
Créée en 2020, la deeptech Qubit Pharmaceuticals s’attelle depuis lors au développement de nouveaux candidats médicaments en misant, pour cela, sur une approche axée autour de trois piliers technologiques complémentaires : le calcul quantique, le calcul hautes performances (HPC) – une technologie hybride qui s’incarne notamment dans son émulateur Hyperion-1 – mais aussi l’intelligence artificielle. Un vaste champ disciplinaire dans lequel la jeune pousse innovante vient justement d’annoncer avoir réalisé une percée majeure, en développant un modèle de fondation aux capacités a priori inédites : FeNNix-Bio1. Sorte de couteau suisse de la modélisation moléculaire, l’outil permettrait en effet de modéliser de manière dynamique le comportement de molécules de tous types, et ce avec une précision mais aussi une rapidité jamais atteintes jusqu’à présent d’après ses créateurs. De quoi ouvrir des perspectives aussi inédites que prometteuses dans le secteur pharmaceutique, mais aussi bien d’autres domaines, comme nous l’expliquent Jean-Philip Piquemal, professeur à Sorbonne Université, directeur du Laboratoire de Chimie Théorique[1], co-fondateur et directeur scientifique de Qubit Pharmaceuticals, et Robert Marino, P.-D.G. de Qubit Pharmaceuticals.
Techniques de l’Ingénieur : Qu’est-ce qui vous a conduits à vous lancer dans le développement de ce modèle de fondation novateur sur lequel vous levez aujourd’hui le voile, FeNNix-Bio1 ?

Jean-Philip Piquemal : L’origine fondamentale du projet tient tout simplement à la raison d’être de Qubit Pharmaceuticals, qui vise en effet avant tout à développer des outils de modélisation moléculaire. Ceci, dans le but de remplacer la phase préclinique du cycle de développement d’un médicament par un ensemble de calculs, et ainsi de réduire drastiquement les besoins en matière de synthèse, et donc, in fine, de permettre des économies de temps, mais aussi d’argent substantielles.
À la création de l’entreprise en 2020, des modèles d’IA dans ce domaine existaient déjà, certes, et se révélaient d’ailleurs assez rapides pour beaucoup d’entre eux, mais n’offraient toutefois pas suffisamment de précision. Or, dans le domaine pharmaceutique, allier rapidité et précision est tout simplement crucial. Cette alliance de la rapidité et d’une précision chimique[2] comparable à celle de l’expérience constitue ainsi le fondement de nos travaux de recherche.
Nous avons donc lancé, il y a quelques années, de premières études dans le domaine de l’IA, avec l’objectif de développer des modèles susceptibles « d’apprendre » la physique et la chimie.
À cela s’est ajouté notre objectif de nourrir ces modèles à partir, non pas de données réelles issues d’expériences menées en laboratoire, mais de données dites « synthétiques », bien moins coûteuses à produire, car fruits, elles aussi, de calculs informatiques.
Nous avons, entretemps, assisté à la naissance d’AlphaFold, un système d’IA capable de prédire la structure 3D des protéines. Cet outil – qui a d’ailleurs valu le prix Nobel de chimie l’an dernier à ses créateurs – a rapidement été présenté comme une avancée susceptible de révolutionner la recherche pharmaceutique. Sauf que, dans ce domaine, la structure des protéines ne fait pas tout, loin de là.
D’où notre idée d’aller au-delà de la seule modélisation de la structure des protéines, en développant un modèle d’IA particulier, car appartenant à la catégorie des « foundation models », ou « modèles de fondation » en français, et permettant de modéliser l’interaction d’un médicament avec sa cible quelle qu’elle soit : protéine, ARN, ADN, membrane cellulaire… Ce qu’AlphaFold n’est pas capable de faire correctement, même dans sa dernière version, AlphaFold 3. Ceci, notamment parce que la structure des protéines n’est pas figée, qu’elle évolue au cours du temps, ce qui implique la réalisation d’une quantité colossale de calculs pour modéliser l’interaction d’un médicament avec cette structure mouvante. Des calculs qui n’étaient, jusqu’à très récemment, réalisés que sur la base de méthodes physiques soit très exactes mais aussi très coûteuses, soit limitées par leur côté « paramétrique », nécessitant des semaines, voire des mois d’ajustements. C’est dans ce contexte qu’a émergé, l’an dernier, le modèle de fondation que nous dévoilons aujourd’hui, FeNNix-Bio1.
Quelles sont les particularités de ce modèle ? Qu’est-ce qui le distingue fondamentalement de ses prédécesseurs ?
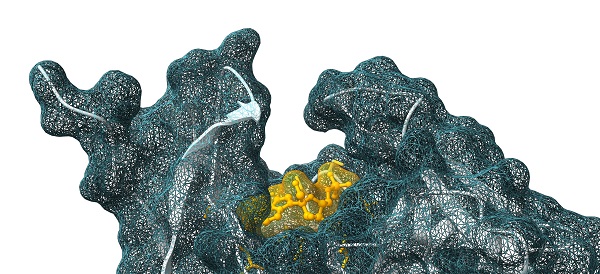
J.-P. P. : Notre modèle de fondation se positionne en aval d’un modèle tel qu’AlphaFold : à partir d’une structure protéique donnée, il se révèle capable d’en simuler la dynamique, mais aussi et surtout les interactions avec une autre substance, notamment une potentielle molécule thérapeutique. Le tout, en un temps très réduit, et avec une précision équivalente à ce que l’on obtient par l’expérience. Et ce, en outre, sans nécessiter la moindre donnée expérimentale d’entraînement. De par notre savoir-faire dans le domaine du calcul hautes performances, nous avons en effet misé sur la production de données synthétiques, un vaste ensemble – des millions – de « briques » élémentaires semblables à des fractions de protéines. Il s’agit, concrètement, de petites molécules composées de deux à cinquante atomes, accompagnées de leur configuration électronique, elle-même modélisée grâce à des calculs basés sur la physique quantique. Produites une fois pour toutes grâce à des supercalculateurs[3], ces données synthétiques nous ont ainsi permis, ensuite, d’entraîner notre modèle de fondation en assemblant tout simplement ces briques à la manière d’un Lego, sans recourir à des données expérimentales. C’est l’une des grandes forces de notre approche.
À partir de cela, notre modèle de fondation se révèle capable de simuler des réactions chimiques de manière dynamique, « remplaçant » ainsi l’équation de Schrödinger, avec un niveau d’erreur très réduit, inférieur à celui de l’expérience. Nous avons d’ailleurs battu un record de précision, que nous présentons dans l’une des deux prépublications[4] que nous avons mises en ligne récemment.

Robert Marino : Quelques-unes de ces données synthétiques ont d’ailleurs été générées grâce à notre émulateur Hyperion-1. Ce n’est qu’un début, mais cela démontre en tout cas, d’une part, que c’est faisable dès maintenant, mais aussi, d’autre part, qu’à partir du moment où nous disposerons d’un véritable ordinateur quantique, nous pourrons encore améliorer et accélérer les choses. Nous anticipons ainsi la convergence entre informatique quantique et IA.
J.-P. P. : Une autre particularité de FeNNix-Bio1 est qu’il « connaît » la réactivité des molécules. Cela va ainsi aider les chimistes médicinaux à identifier des voies de rétrosynthèse. Une fois modélisée, il faut en effet qu’une molécule thérapeutique puisse, concrètement, être synthétisée en laboratoire. Il s’agit là aussi d’une avancée majeure par rapport aux modèles existants jusqu’alors.
À quelles applications concrètes ce modèle de fondation se destine-t-il ?
J.-P. P. : Puisqu’il ne se limite pas à la modélisation de structures, mais qu’il permet aussi de simuler des réactions chimiques, FeNNix-Bio1 se destine à n’importe quel type d’application en chimie, qu’elle soit pharmaceutique ou non. Notre modèle de fondation est par exemple également capable de produire, en quelques secondes, des données telles que des spectres RMN, infrarouges, UV, etc. Il regroupe ainsi toute la panoplie des outils du chimiste.
Parce qu’il est nourri de données issues de la physique quantique, FeNNix-Bio1 permet aussi de simuler le comportement physique de l’eau dans ses différentes phases, et de modéliser ainsi avec précision le comportement des ions et des molécules en solution. Il s’agit d’une avancée majeure, dont ne sont absolument pas capables les autres modèles, tels qu’AlphaFold.
Cela se révèle notamment crucial en chimie pharmaceutique, puisque toutes les cibles potentielles des médicaments baignent dans l’eau salée dont est en grande partie constitué le corps humain. On peut ainsi modéliser des interactions de manière dynamique, comme si l’on filmait ce qui se passe réellement dans nos tissus, et ce, à une vitesse compatible avec des travaux de recherche pharmaceutique.
La palette d’applications potentielles est d’autant plus large que notre modèle peut être réentraîné sur de nouvelles données en l’espace de quelques jours seulement, là où les modèles de langage comme ceux de ChatGPT, par exemple, nécessitent des mois – et donc aussi de grandes quantités d’énergie – pour évoluer.
R.M. : Nous nous concentrons, dans le cadre de nos travaux de développement de nouveaux candidats médicaments, sur différents axes dans le domaine pharmaceutique, mais notre modèle – parce qu’il peut facilement être réentraîné sur n’importe quel type de données comme vient de l’indiquer Jean-Philip – va aussi trouver des applications dans tous les domaines de la chimie et des sciences de la vie en général : en chimie pharmaceutique et cosmétique, mais aussi dans le domaine des enzymes industrielles, ou encore du côté de la chimie des matériaux, pour produire par exemple des électrolytes de batteries électriques, ou encore des membranes de dessalement.
L’an dernier, la jeune pousse Bioptimus[5] s’est elle aussi lancée dans un projet de développement d’un modèle de fondation dédié à l’étude du vivant. Faut-il y voir un potentiel concurrent de votre modèle, FeNNix-Bio1 ?
R.M. : Il s’agit plutôt d’une solution complémentaire à la nôtre. Leur modèle, h-optimus-1, se destine en effet à la compréhension de mécanismes biologiques qui entrent en jeu dans le cadre d’une maladie, de l’échelle moléculaire jusqu’au niveau d’un organisme tout entier. Notre modèle FeNNix-Bio1, lui, va de l’électron à la molécule. Les modèles de fondation sont, de toute façon, amenés, à terme, à interagir les uns avec les autres : AlphaFold peut d’ores et déjà générer des données utilisables par FeNNix-Bio1, qui lui-même pourra tout à fait transmettre des résultats au modèle de Bioptimus. Chacun travaille à son échelle, avec des données différentes.
Outre son utilisation pour vos propres travaux de développement de candidats médicaments, comptez-vous justement mettre à disposition votre modèle FeNNix-Bio1 à d’autres acteurs ? Si c’est le cas, selon quelles modalités ?
R.M. : Nous allons effectivement, bien entendu, exploiter ce modèle pour découvrir de nouveaux médicaments. Mais notre objectif est aussi qu’il soit utilisé plus largement par d’autres acteurs de la recherche académique et industrielle. Cela nous semble en effet d’autant plus important que le développement de FeNNix-Bio1 est le fruit d’un partenariat public-privé entre Sorbonne Université et Qubit Pharmaceuticals.
J.-P. P. : Nous allons en effet mettre à disposition notre modèle à la fois dans une déclinaison open-source[6], mais aussi dans une version propriétaire destinée à des applications industrielles plus poussées. Grâce à la possibilité de réentraînement qui le caractérise, c’est même à toute une famille de modèles – plus spécialisés – que FeNNix-Bio1 permettra de donner naissance ; publics pour certains d’entre eux, propriétaires pour d’autres. Ce modèle de fondation est ainsi un véritable couteau suisse, qui va permettre de générer beaucoup d’autres solutions, qui viendront non pas remplacer, mais plutôt « augmenter » les chimistes humains.
[1] Sorbonne Université/CNRS
[2] Seuil fixé à 1 kcal/mol, en dessous duquel le résultat est indiscernable de l’expérience.
[3] Supercalculateurs Jean Zay, EuroHPC et Aurora.
[4] Plé T., et al. A Foundation Model for Accurate Atomistic Simulations in Drug Design. ChemRxiv. 2025 ; Benali A., et al. Pushing the Accuracy Limit of Foundation Neural Network Models with Quantum Monte Carlo Forces and Path Integrals. arXiv. 2025.
[6] Le code est pour l’heure accessible aux relecteurs des prépublications actuellement soumises à des revues à comité de lecture par Qubit Pharmaceuticals. Une partie de ce code sera également rendue publique cet été.





















Réagissez à cet article
Vous avez déjà un compte ? Connectez-vous et retrouvez plus tard tous vos commentaires dans votre espace personnel.
Inscrivez-vous !
Vous n'avez pas encore de compte ?
CRÉER UN COMPTE