Faire pousser des tissus et des organes sur des systèmes microfluidiques miniatures, c’est à cela que se résument les organes sur puce. Sur ces surfaces qui font tout au plus un millimètre, sont ainsi mimées les fonctions physiologiques. Comment sont-elles construites et quelles en sont les applications ?
Discipline pluridisciplinaire par excellence, à mi-chemin entre la biologie et l’ingénierie, les organes sur puce ont révolutionné la culture cellulaire. Sur une puce gravée d’un système microfluidique (qui consiste en des canaux traversés par un liquide physiologique), des cellules sont ensemencées pour ensuite former des tissus voire même des organes. Il est même possible de connecter des puces entre elles pour reproduire le fonctionnement de l’organisme. « La taille d’une puce fait entre 100 micromètres et un millimètre, nous sommes donc au stade des micromachines et de la microtechnologie », souligne Dominique Collard, directeur de recherche au LIMMS, un laboratoire spécialisé dans la recherche sur les microsystèmes, commun entre le CNRS et l’Université de Tokyo (Japon).
Fabrice Soncin, qui dirige une équipe de recherche intégrée au projet SMMiL-E, un programme tourné vers la cancérologie, également au LIMMS, explique le concept : « Tout comme un organe est constitué d’une apposition de différents tissus formés de divers types cellulaires, les organes sur puce miment cela in vitro. Le grand avantage est que, grâce aux systèmes microfluidiques, nous parvenons à perfuser les cellules avec leur milieu de culture. » Directeur du laboratoire Biologie à grande échelle (CEA, Inserm et Université Grenoble-Alpes), Xavier Gidrol renchérit : « Dans un système microfluidique, le milieu de culture circule en permanence, ce qui permet de garder les cellules vivantes pendant plusieurs mois, voire même une année ».
Une micro-fabrication par photolithographie
L’approche de microfabrication est résumée par Fabrice Soncin : « Au départ, on grave des galettes de silicium : on construit à très haute résolution un moule inverse de ce qu’on veut et, sur ces moules, on fait couler du PDMS, un matériau biocompatible qui va se solidifier pour former des microcanaux de l’ordre de quelques micromètres, soit une taille manipulable et visualisable au microscope. C’est dans ces microcanaux que circulera le milieu de culture et qu’on ensemencera les cellules ».
Cagatay Tarhan, diplômé de l’Université de Tokyo et enseignant-chercheur chez Yncréa Hauts de France et au LIMMS, ajoute : « Avant le moulage avec le PDMS, nous avons recours à la photolithographie, mais à l’échelle du micron. Son fonctionnement rappelle celui de la photographie. »
On vous explique la démarche photolithographique dans le dictionnaire technique multilingue de Techniques de l’Ingénieur : « Une fine couche de résine photosensible est tout d’abord déposée sur le substrat. Elle est ensuite exposée à une source lumineuse (rayonnement ultra-violet UV) à travers un masque optique présentant des zones opaques et transparentes et reproduisant les motifs planaires du circuit à graver. Les motifs sont ensuite révélés dans une solution chimique, ce qui aboutit à une reproduction des motifs du masque en négatif ou en positif selon le type de résine utilisé. »
Reproduire des vaisseaux sanguins tumoraux
L’équipe de Fabrice Soncin travaille à fabriquer des modèles 3D in vitro de vaisseaux sanguins, l’angiogenèse (formation de nouveaux vaisseaux sanguins) étant l’un des mécanismes essentiels de la croissance tumorale. « Cela nous permet de tester in vitro des molécules médicamenteuses et de limiter fortement l’utilisation de modèles animaux », souligne le directeur de recherche de l’Inserm.
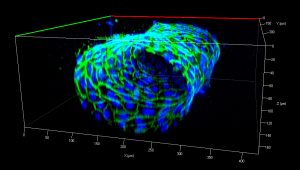
Etape par étape, il relate comment reproduire des vaisseaux sanguins perfusés : « Nous gravons des galettes de silicium pour y créer le moule à l’inverse et nous faisons couler dedans le PDMS qui, en se polymérisant, va devenir comme du plastique poreux. Une fois que ces canaux sont créés, nous les collons sur une lame de verre pour fermer l’une des faces des canaux de manière à ce que ce soit hermétique. Ensuite, nous connectons les canaux à un système fluidique : contrôleurs de pression, débitmètres, valves, ou autre… Concrètement, nous faisons geler dans ces canaux un hydrogel qui servira de matrice extracellulaire sur laquelle les cellules s’établiront. À l’intérieur de cet hydrogel, nous créons un premier canal en continuité avec le système de perfusion, et c’est à travers ce système fluidique que nous ensemençons les cellules endothéliales. Au bout de trois jours, ces cellules forment une monocouche qui vient tapisser le canal. Les cellules endothéliales poussent jusqu’à confluence et s’arrêtent, comme dans le cas d’un vaisseau in vivo. Dans ce canal de cellules endothéliales, nous faisons passer du milieu de culture dans des conditions qui miment la perfusion sanguine, et des cellules circulantes de l’immunité, qui participent aux réactions inflammatoires ».
Toujours selon le chercheur, différentes analyses peuvent être menées par la suite sur cette structure in vitro : l’observation en temps réel sous microscope confocal (en immunofluorescence), l’analyse de protéines (par Western Blot), un profilage de l’expression des gènes… Cependant, quand il ne s’agit pas d’observation au microscope, il faut récupérer les cellules : « Pour ce faire, il est nécessaire de détruire l’organe, précise Fabrice Soncin, par lyse de l’hydrogel ou digestion enzymatique. Or, un problème est que nous avons peu de cellules car nous travaillons sur des dimensions très petites… Pour étudier les protéines, nous avons besoin de beaucoup de cellules et donc de plusieurs puces ». Le chercheur affirme par ailleurs qu’un même moule peut servir plusieurs fois, « tant qu’il est bon », et servir à créer « une dizaine d’unités de puces », mais cela reste à l’échelle du laboratoire, le but n’étant pas d’en industrialiser la fabrication.
Il existe cependant des entreprises qui commercialisent des puces destinées à la culture cellulaire. C’est le cas de la startup américaine Emulate, dont la technologie est notamment utilisée à l’Institut Pasteur.
Interested in learning how our Lung-Chip could be used for COVID-19 research? Join us on May 7th at 11 am EDT for a webinar on how the Lung-Chip can be used to study viral infection.#research #biotech #organchips #organsonchips #COVID19
— Emulate, Inc. (@emulateinc) April 24, 2020
Cette société est une « spin-out » qui a vu le jour au Wyss Institute, à Harvard (Etats-Unis), le laboratoire précurseur des organes sur puce et à l’origine du premier poumon sur puce.
Organ Chips from the @wyssinstitute and @emulateinc are hopping the pond to be included in the @BarbicanCentre’s “AI: More Than Human” exhibit, which asks the “big questions” like, What does it mean to be human? And will machines ever outsmart a human?https://t.co/16ASEZwZcy pic.twitter.com/daJVcZqCO5
— Wyss Institute (@wyssinstitute) May 16, 2019
Le pancréas sur puce
Ce ne sont pas des vaisseaux sanguins mais un pancréas qui est construit sur puce par l’équipe de recherche de Xavier Gidrol, en collaboration avec l’équipe de Fabrice Navarro au CEA Leti. Le pancréas, comme le rappelle le chercheur, remplit deux fonctions. Une fonction dite « endocrine » : il sécrète l’insuline et le glucagon, des hormones qui régulent le taux de glucose dans le sang. Et une fonction « exocrine » dont la vocation est de sécréter des sucs digestifs qui se déversent dans le côlon pour aider à la digestion.
« Nous travaillons sur plusieurs organes sur puce qui miment l’une ou l’autre de ces fonctions, indique Xavier Gidrol. Nous réalisons des organes sur puce autour des maladies affectant le pancréas, comme le diabète ou le cancer. Le premier modèle que nous avons conçu était des îlots de Langerhans [amas de cellules pancréatiques productrices d’hormones, NDLR]. Le but a été de les vasculariser grâce à des co-cultures afin de permettre aux îlots de sécréter de l’insuline pendant un mois, et nous permettre ainsi de tester des médicaments. En effet, un problème majeur aujourd’hui est l’attrition [également appelée ‘taux d’abandon’, elle correspond à la perte de participants dans un essai clinique, NDLR], et une de ses raisons est l’inadéquation des modèles animaux qui ne sont pas toujours pertinents d’un point de vue scientifique ou bien acceptés d’un point de vue éthique. Les organes sur puce comblent ce vide car ils constituent des modèles plus pertinents pour les tests médicamenteux, en espérant qu’on obtiendrait ainsi moins de rejets et de déchets. »
Est-il possible de mettre au point des substituts d’#organes ? Quelle est la différence entre un #organe sur puce et un #organoïde ? « Mimer le vivant », un article à découvrir dans le dernier numéro des #ClefsCEA sur la #MédecineduFutur
➡ https://t.co/OsWUhTU7YH pic.twitter.com/b02vt1PUI3— CEA_Officiel (@CEA_Officiel) January 16, 2019
Le deuxième modèle de pancréas sur puce conçu dans ce laboratoire vise à déterminer si le diabète augmente ou pas le risque de cancer du pancréas : « La littérature est essentiellement basée sur l’épidémiologie, note Xavier Gidrol, et très peu de papiers essaient d’identifier les mécanismes moléculaires impliqués. Ce que l’on étudie cette fois est la fonction exocrine, en créant des organes sur puce de tissus grâce à des cellules prélevées de personnes non diabétiques et d’autres de patients diabétiques. Nous mimons ainsi le micro-environnement d’un patient diabétique et celui d’un donneur non-diabétique afin d’étudier les conséquences du diabète sur la cancérisation du pancréas, celle-ci émergeant dans la partie exocrine de l’organe ».
Dans cette même logique de recréation de micro-environnement de diabétique et de non-diabétique, l’équipe développe une peau sur puce : « Nous cherchons à savoir comment le diabète influence la cicatrisation de la peau, poursuit Xavier Gidrol. Nous créons artificiellement une blessure et nous étudions comment le micro-environnement diabétique retarde la cicatrisation de la blessure. Nous travaillons sur des cellules de peau, différenciées à partir des cellules humaines d’un donneur, que nous vascularisons en co-culture ».
À la place du PMDS, cette équipe de recherche utilise du COC. « Au début de la discipline, le PDMS était l’élastomère le plus utilisé, explique Xavier Gidrol. Mais il est controversé, car beaucoup de molécules pharmaceutiques s’y accrochent. Le choix de l’élastomère est en fonction de la physiologie que l’on souhaite mimer. Historiquement, le premier organe sur puce était un poumon, et pour celui-ci le PMDS est idéal car il est élastique. Le COC, quant à lui, est rigide. »
Adapter le liquide physiologique, un défi
Pour Fabrice Soncin, la composition de la matrice extracellulaire, le substrat sur lequel les cellules s’accrochent et qui est nécessaire à maintenir les cellules en vie, peut se révéler problématique : « Le souci est dans la composition et la reproductibilité. Cette matrice est extraite de tissus vivants et comme ce sont des matériaux biologiques, leur composition diffère entre les lots achetés. Elle peut être riche en collagène issu de tendons d’animaux mais il existe aussi des hydrogels plus spécifiquement conçus pour la culture de cellules tumorales mais leur composition n’est pas non plus connue et varie aussi en fonction des lots achetés. Nous nous retrouvons à nous poser des questions comme : est-ce que nos observations sont finalement dues à la composition spécifique du lot ? L’idéal est de créer une matrice de synthèse pour contrôler beaucoup mieux la composition et la structure. En ce sens, nous avons démarré un partenariat avec une entreprise de biotechnologie qui commercialise une matrice synthétique de composition et de fabrication connues ».
Une recherche en synergie
Des scientifiques aux profils variés interviennent dans la recherche sur les organes sur puce. Leurs expertises se complètent. « Je suis ingénieur spécialisé en électronique, témoigne Cagatay Tarhan, directeur du programme SMMiL-E, mais l’équipe comprend également des biologistes, des ingénieurs en mécanique, des biophysiciens, des scientifiques spécialistes des matériaux, des chimistes, des data scientists, des statisticiens… ». L’esprit collaboratif ne s’arrête pas là et dépasse même les frontières françaises, comme l’explique Dominique Collard : « Le LIMMS a été fondé il y a 25 ans et le but à la base était de développer de nouveaux microsystèmes. Ensuite, une fois que cette technologie est devenue suffisamment mature, cela nous a permis de l’appliquer à la médecine et à la biologie. Nous avons alors entrepris de transférer la technologie du Japon jusqu’en France où nous pouvons travailler sur des études précliniques. Et c’est à Lille que l’opportunité s’est présentée pour mener des travaux en cancérologie avec le Centre Oscar Lambret et en lien avec l’Université de Lille ». Des chercheurs japonais participent régulièrement à des travaux collaboratifs en se rendant sur place, au LIMMS lillois.
Flashback on the SYMPOSIUM last Thursday @ipggmicrofluid celebrating 20th ANNIVERSARY of Prof. Teruo FUJII’s LAB at #IIS as well as the🇫🇷🇯🇵@limmsTOKYO Int Research Lab of @CNRS & @UTokyo_News_en with nice photographs taken by Anthony Genot (thank you Anthony!) #CNRSInternational pic.twitter.com/9cNY7GLsds
— CNRSinJapan (@CNRSinJapan) October 28, 2019
Pour sa part, l’équipe de Xavier Gidrol s’implique dans plusieurs projets européens : elle fait notamment partie de la European Organ-on-Chip Society (EUROoCS). En 2018, année de sa création, l’EUROoCS a publié un projet de roadmap pour la recherche européenne future portant sur les organes sur puce. « Tous les ans, l’EUROoCS organise un colloque où se réunissent ses membres », indique Xavier Gidrol. En raison de la pandémie de Covid-19, l’édition 2020 du colloque annuel s’est déroulée en ligne, en juillet. Il se tiendra à Uppsala (Suède) en 2021.
Image de une : Puce microfluidique à visée médicale. Crédit photo : FMNLab / CC BY (https://creativecommons.org/licenses/by/4.0)





















Réagissez à cet article
Vous avez déjà un compte ? Connectez-vous et retrouvez plus tard tous vos commentaires dans votre espace personnel.
Inscrivez-vous !
Vous n'avez pas encore de compte ?
CRÉER UN COMPTE